Uitheemse rivierkreeften worden maar beperkt structureel gemonitord. Onderzocht is of met eDNA technieken ook een eenvoudiger uit te voeren kwantificering van het aantal rode Amerikaanse rivierkreeften gemaakt kan worden.
Kwantitatieve eDNA-bemonstering ten behoeve van rode Amerikaanse rivierkreeften
Uitheemse rivierkreeften worden maar beperkt structureel gemonitord. In 2010 en 2019 is landelijk een verspreidingsonderzoek georganiseerd, maar dit heeft een betrekkelijk lage dichtheid aan meetpunten. Incidenteel wordt in een gebied de kreeftendichtheid in kaart gebracht. Een probleem dat een regulier meetprogramma in de weg staat is de complexiteit van het organiseren daarvan: voor iedere uitgezette fuik moeten de juiste vergunningen en toestemmingen worden verkregen, en per opname moeten tweemaal de locaties bezocht worden met het risico van tussentijds verdwijnen van de fuik. Om een nieuw, eenvoudiger in te zetten middel in handen te krijgen, is onderzocht of met eDNA technieken ook een kwantificering van het aantal rode Amerikaanse rivierkreeften gemaakt kan worden.
Problemen van uitheemse rivierkreeften
Met de opkomst van de Amerikaanse rivierkreeften in Nederland worden tal van problemen ervaren. Die problemen lopen uiteen van knip- en vraatschade aan vegetatie en predatie op en competitie met fauna, tot graafschade aan oevers en verminderde vangsten voor sportvissers. Om grip te krijgen op deze problemen, worden momenteel allerlei onderzoeken georganiseerd en uitgevoerd. Een terugkerend aspect bij dergelijke onderzoeken is dat wanneer aantallen of dichtheden van kreeften moeten worden bepaald, er fuiken en korven nodig zijn om de kreeften te kunnen tellen. Dergelijke vangmiddelen zijn als vistuig in de visserijwetgeving opgenomen. Dit betekent dat er voor gedegen onderzoek altijd een vergunning en/of hulp van een beroepsvisser nodig is. Daarnaast is toestemming van de visrechthebbende van het te bevissen water nodig, en deze toestemming is niet altijd makkelijk te krijgen. Dit compliceert het opzetten van onderzoek, zeker als het gaat om de grootschalige en terugkerende inspanning van reguliere monitoring. Een inventarisatiemethode die niet onder de visserij-wetgeving valt kan dus veel opleveren.
Rivierkreeften en eDNA, een mooie kans voor onderzoek
Door ATKB, Witteveen+Bos en Datura is, in opdracht van Waternet en de VBNE, op een aantal locaties een kleinschalig onderzoek uitgevoerd naar rivierkreeften en eDNA. Uit deze verkenning kwam naar voren dat er een mogelijk bruikbaar verband te vinden is tussen de aantallen rivierkreeften en de hoeveelheid eDNA in het water. Een dergelijke techniek kan een eenvoudiger vorm van rivierkreeftenmonitoring betekenen, als dit verband wordt bevestigd.
In 2019 is onder leiding van de STOWA een grootschalig onderzoek naar rivierkreeften georganiseerd. Hierin is gezocht naar correlaties tussen de aantallen rode Amerikaanse rivierkreeften en allerlei omgevingsfactoren. Het doel was het zoeken naar mogelijke stuurfacto-ren die invloed hebben op die aantallen. Voor dit onderzoek zijn een groot aantal locaties bezocht, waar de aantallen kreeften in kaart zijn gebracht met behulp van fuiken. Dit was een interessante kans om het kleinschalige eDNA onderzoek van de benodigde schaalvergroting te voorzien. De kosten konden zo aanzienlijk worden beperkt doordat het benodigde fuikenonderzoek al werd georganiseerd.
Deze kans is met beide handen aangegrepen. Met behulp van financiering uit het Innovatie-fonds van het Hoogheemraadschap van Delfland is het eDNA onderzoek opgezet. Het bemonsteren en analyseren van eDNA van rivierkreeften, gelijktijdig met het uitzetten van fuiken voor het STOWA onderzoek, is door Witteveen+Bos en Datura in gang gezet.

Fuikbemonstering Foto: Tessa van der Wijngaard
Opzet van het onderzoek
Er zijn op 75 locaties, verdeeld over de deelnemende waterschappen, watermonsters voor eDNA-analyse genomen. Op iedere locatie is 1 mengmonster van 1 liter water verzameld. Tegelijkertijd is het aantal kreeften bepaald door 12 beaasde fuiken 1 nacht uit te zetten.
De analyse is met 5 eDNA-analysemethodes uitgevoerd: 4 gebaseerd op qPCR, en 1 met ddPCR. Het principe van de methodes is dat een voor de rode Amerikaanse rivierkreeft uniek stukje DNA via herhaalde verdubbeling wordt verveelvoudigd. Dit wordt net zo lang gedaan tot de concentratie DNA een drempelwaarde overschrijdt. Methode 1 t/m 4 zijn uitgevoerd via qPCR. Methode 1, het aantal benodigde verdubbelingen ofwel de Ct-waarde, is hierbij een indicatie voor de concentratie. Een omrekening naar aantal moleculen met en zonder correctie voor verstorende stoffen, vormt methode 2 en 3. Het aantal positieve replica’s methode 4. Het resultaat van methode 1 kan niet worden vergeleken met het resultaat van een andere qPCR machine. Het resultaat van methode 2 t/m 4 wel, maar deze kunnen veel ruis introduceren. Daarom is gedurende het onderzoek de 5e methode, ddPCR, geïntroduceerd. Deze is vergelijkbaar met qPCR, maar het monster wordt nu verdeeld over duizenden oliedruppeltjes. Hier komt direct een concentratie uit, waardoor het resultaat tussen verschillende machines vergelijkbaar is zonder ruis-introducerende correcties. Voor een deel van de monsters was helaas te weinig materiaal over voor deze aanvulling.
Gevonden verbanden
Er is een verband gevonden tussen de aantallen gevangen rivierkreeften en het aanwezige eDNA. Het sterkste verband bleek de Ct-waarde van de qPCR te geven. Die was bijna 38% verklarend voor het aantal rivierkreeften (R2-waarde van 0.3771). Methodes 2 t/m 4 waren (veel) zwakker doordat veel ruis ontstond bij de bepaling daarvan. Bij de ddPCR werd een verband gevonden dat bijna 26% van het aantal gevangen rivierkreeften kon verklaren (R2-waarde van 0.2562).
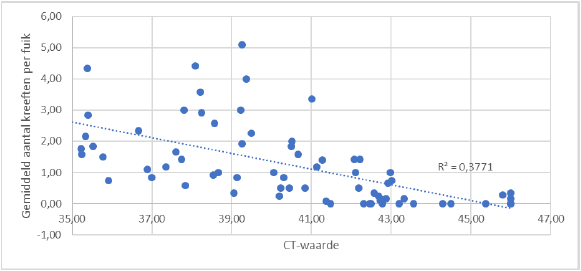
Het sterkste gevonden verband; De relatie tussen het aantal temperatuurcycli dat nodig is om de hoeveelheid eDNA de drempelwaarde voor detectie te laten passeren (Ct-waarde) en het gemiddelde aantal kreeften per fuik.
Een vervolg
Het verband tussen eDNA en de aas-fuikmethode is nu niet zo sterk dat gelijk een meetprotocol kan worden uitgewerkt. Echter, het is de verwachting dat de eDNA-methoden verder geoptimaliseerd kunnen worden, met name de ddPCR-methode. Daarom is het nu de verwachting dat bepaling van de aantallen rivierkreeften met eDNA tot de mogelijkheden kan behoren. Zodoende wordt in de komende tijd verkent wat de mogelijkheden van een verdere optimalisatie zijn.
Geïnteresseerden zijn zeker welkom om contact op te nemen met de auteur van dit artikel (
Tekst: Ernst Raaphorst, Hoogheemraadschap van Delfland
Verschenen in de nieuwsbrief plaagsoorten juni 2020

